La bronchite infettiva (BI) è una patologia virale che può manifestarsi con forme respiratorie, renali o riproduttive in polli di tutte le età e categorie produttive, causando danni ingenti alla produzione avicola. Il suo agente eziologico, noto come virus della bronchite infettiva (IBV), è endemico a tutte le latitudini, obbligando a un ricorso pressoché routinario alla vaccinazione per garantire un’adeguata protezione. Essa è principalmente basata sull’utilizzo di vaccini vivi attenuati e secondariamente sulla somministrazione di vaccini inattivati per il boosting immunitario di ovaiole e riproduttori.
Trattandosi di un virus a RNA appartenente alla famiglia dei coronavirus, IBV è contraddistinto da un alto tasso di mutazione, che, unito a un’elevata propensione alla ricombinazione, causa il continuo emergere di nuove varianti genetiche, caratterizzate da differenze anche significative in termini di manifestazione clinica, capacità di indurre protezione reciproca e distribuzione geografica. Questa variabilità ha profonde implicazioni pratiche sia in termini di controllo, richiedendo spesso la somministrazione di più vaccini basati su ceppi diversi, sia in termini di diagnosi, la quale deve essere intrapresa con test in grado di rilevare le varianti di interesse e che richiede un aggiornamento costante.
La classificazione genetica di IBV attualmente in uso si basa sul sequenziamento del gene codificante per la subunità S1 della proteina Spike, posta sulla superficie dei Coronavirus e che per questo svolge un ruolo essenziale nell’evocare la risposta immunitaria e nel determinare quali specie e tessuti saranno suscettibili all’infezione. Ad oggi, sono noti otto diversi genotipi di IBV (GI-GVIII), i quali possono essere ulteriormente suddivisi in più di trenta lineage (GI-1, GI-2, G2-1, ecc.).
Una delle novità epidemiologiche più rilevanti degli ultimi anni è la descrizione del ceppo denominato IB80, principale esponente del lineage GVIII-2. A partire dal 2015, la presenza di IB80 è stata documentata in numerosi paesi europei (Polonia, Belgio, Germania, Paesi Bassi, Spagna, Ucraina, Lituania, Bielorussia, Russia) oltre che in Kazakistan, Giordania, Arabia Saudita e Filippine. Tali ritrovamenti sono avvenuti principalmente in galline ovaiole e riproduttori, con una possibile associazione a cali dell’ovodeposizione sia in termini qualitativi che quantitativi. Ciononostante, l’effettivo impatto clinico di IB80 risulta tuttora poco chiaro, in quanto nel corso dell’unico test di patogenicità svolto sinora con un isolato appartenente al lineage GVIII-2 non sono stati osservati sintomi né respiratori né riproduttivi.
A causa dell’elevata diversità genetica rispetto al resto dei ceppi di IBV circolanti, molte delle metodiche di biologia molecolare comunemente utilizzate per la diagnosi di IBV non consentono la rilevazione di IB80. Ciò ha complicato l’esecuzione di approfondimenti epidemiologici, impedendo l’accertamento della presenza (o assenza) di IB80 in molti paesi, inclusa l’Italia.
La ricerca di IB80 sul territorio italiano ha quindi richiesto dapprima la validazione e l’implementazione di una metodica di biologia molecolare specifica per questo ceppo. Tramite analisi bioinformatiche condotte sulle sequenze del gene S1 di ceppi GVIII disponibili in database pubblici, sono state messe a punto diverse soluzioni diagnostiche basate sulla reazione a catena della polimerasi con trascrittasi inversa (RT-PCR), che consente la rilevazione del materiale genetico a cui può seguire il suo sequenziamento. Si è quindi proceduto con l’individuazione della metodica più performante attraverso la conduzione di prove svolte con l’utilizzo di un plasmide, una piccola molecola di DNA circolare, nel quale è stata inserita la sequenza del gene S1 del ceppo IB80. Tali prove hanno poi consentito di affinare la metodica per ottimizzarne le prestazioni in termini di specificità, sensibilità e ripetibilità. Il test risultante si è dimostrato essere in grado di rilevare i ceppi appartenenti a GVIII fino a una concentrazione minima di 10 copie/µL, evitando al contempo possibili false positività date dalla presenza nel campione di ceppi appartenenti ad altri genotipi di IBV di comune ritrovamento o di altri patogeni respiratori, come il virus della malattia di Newcastle (NDV), il metapneumovirus aviare (aMPV) e il virus della laringotracheite infettiva (ILTV).
Successivamente alla validazione, il test specifico per GVIII è stato utilizzato nell’ambito delle attività di ricerca svolte presso il Laboratorio di Microbiologia e Malattie Infettive del Dipartimento MAPS dell’Università di Padova come parte di un pannello di metodiche per la ricerca di IBV selezionate sulla base del contesto epidemiologico e delle vaccinazioni adottate. Alla luce della maggior frequenza di ritrovamento di IB80 in tipologie produttive a lunga vita, la metodica validata è stata utilizzata per analizzare i campioni ricevuti da allevamenti di ovaiole e riproduttori, che consistevano in tamponi prelevati a livello tracheale o cloacale.
In caso di risultato positivo, i campioni sono stati sottoposti a sequenziamento della porzione del genoma amplificata, la quale ha poi permesso la caratterizzazione del virus tramite analisi filogenetiche. Oltre alla metodica GVIII-specifica sono state condotte ulteriori analisi con una metodica di RT-PCR generica, che consentiva la rilevazione delle varianti di IBV di comune ritrovamento e ha quindi permesso l’individuazione di eventuali coinfezioni.
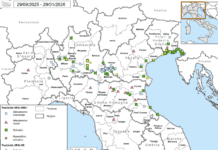
Tra l’aprile e il giugno 2024, sono stati analizzati campioni prelevati da 84 capannoni appartenenti a 24 diversi allevamenti. In totale, la metodica ha evidenziato positività in 11 capannoni (13%) di 5 allevamenti diversi (20%) (Figura 1).
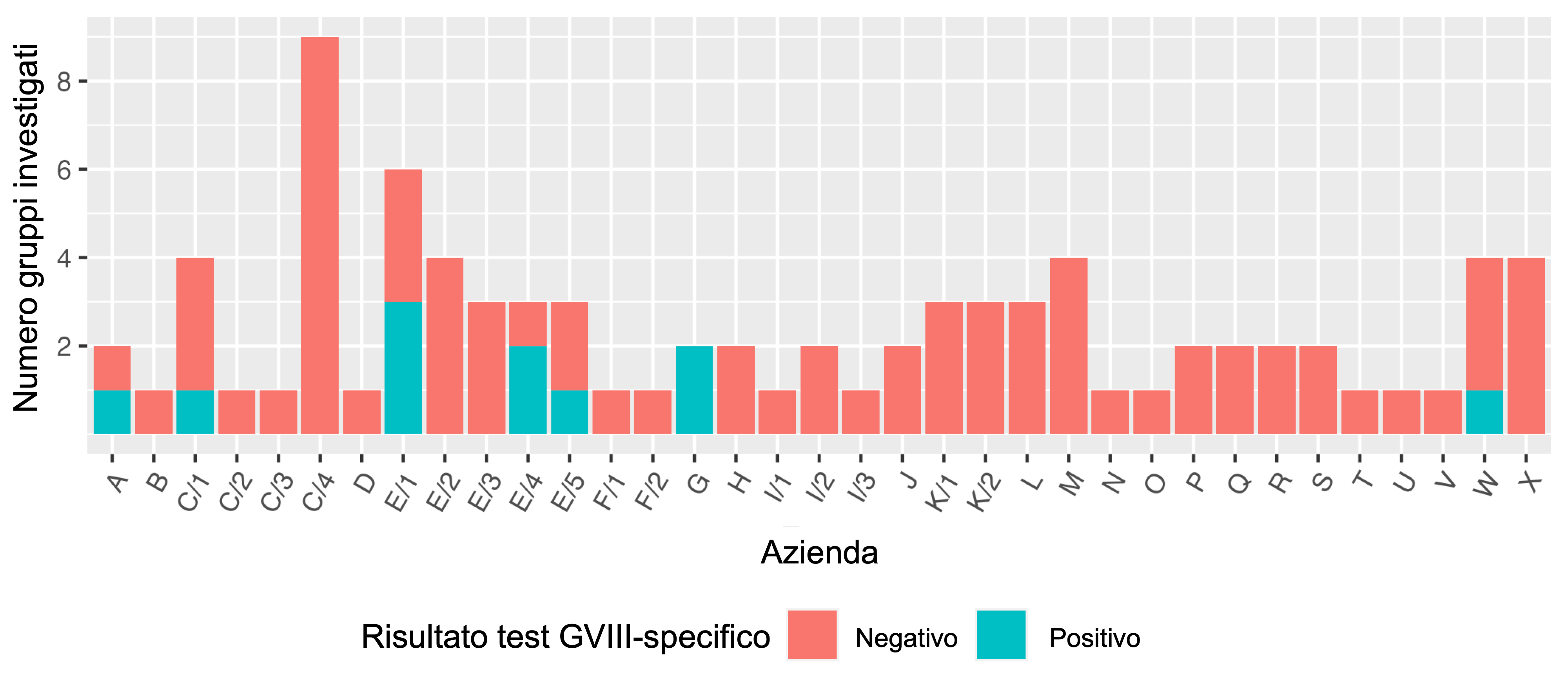
Gli allevamenti di ovaiole positivi erano localizzati in Veneto, Lombardia, Friuli Venezia-Giulia ed Emilia-Romagna, mentre nessuna positività è stata rilevata in Lazio e Piemonte. Utilizzando la metodica di RT-PCR generica sono state invece accertate 18 positività (21,4%) in 12 allevamenti (50%) a ceppi appartenenti al lineage
GI-13 (793/B) e di origine vaccinale, oltre a 7 positività (8,3%) in 3 allevamenti (12,5%) a virus di campo appartenenti al lineage GI-19 (QX). Sono state rilevate 5 coinfezioni, 2 tra ceppi GVIII e 793/B e 3 tra ceppi GVIII e QX (Tabella 1).
Le positività alla metodica GVIII-specifica sono risultate ascrivibili a ceppi con un’identità genetica reciproca compresa tra il 96,8% e il 99,7% a livello di gene S1. La comparazione con le altre sequenze di ceppi GVIII (Figura 2) ha permesso di determinare l’appartenenza delle sequenze italiane al lineage GVIII-2 e, in particolare, a una clade che comprendeva anche un ceppo identificato in Giordania nel 2021. L’identità genetica con il ceppo IB80, appartenente a una clade contenente solo sequenze tedesche, era invece compresa tra il 96,8% e il 97,7%.

I risultati ottenuti hanno quindi permesso di confermare la presenza di ceppi GVIII-2 in Italia. Nonostante si tratti della prima segnalazione in tal senso, tale novità epidemiologica è probabilmente da ascrivere al mancato (o limitato) utilizzo di metodiche in grado di rilevarli piuttosto che a una loro recente introduzione nel nostro territorio. La circolazione di tale lineage è apparsa significativa, interessando circa il 20% degli allevamenti campionati. In alcuni di essi le positività sono inoltre avvenute in gruppi e momenti di campionamento differenti, dimostrando la possibilità di una prolungata circolazione interna. I virus identificati hanno mostrato un’elevata identità genetica, venendo raggruppati separatamente da ceppi dello stesso lineage ritrovati in altre parti d’Europa. Curiosamente, la sequenza genetica più vicina è risultata provenire dalla Giordania, sebbene il basso numero di sequenze disponibili pubblicamente limiti le conclusioni epidemiologiche che si possono trarre dalla filogenesi.
Confermando quanto già segnalato nella letteratura attualmente disponibile, i ritrovamenti di GVIII-2 sono avvenuti in gruppi di ovaiole, ma la possibilità che questi ceppi circolino anche in altri comparti produttivi risulta tuttora da indagare. Ulteriori interrogativi riguardano l’effettiva rilevanza clinica di tali infezioni, in quanto l’anamnesi relativa ai gruppi positivi è risultata spesso incospicua e comunque non univoca, con un solo gruppo interessato da problemi respiratori e altri due (appartenenti però allo stesso allevamento) da cali dell’ovodeposizione. Conseguentemente, non è stato possibile confermare l’effettivo impatto dei ceppi identificati, la cui definizione richiederà studi dedicati.
La segnalazione della presenza di ceppi GVIII-2 in Italia risulta di grande importanza pratica, in quanto evidenzia la necessità di aggiornare gli approcci diagnostici attualmente utilizzati nei confronti di IBV a livello nazionale. Solo col ricorso a metodiche specifiche risulterà infatti possibile confermare la presenza (così come l’assenza) di tale lineage nell’ambito della diagnostica routinaria. A fronte delle limitate soluzioni diagnostiche a oggi disponibili, la validazione di una metodica di RT-PCR di facile applicabilità va quindi considerata un risultato egualmente rilevante rispetto alla produzione di dati epidemiologici, offrendo un’utile risorsa per la ricerca dell’intero genotipo GVIII ed essendo quindi potenzialmente utilizzabile anche in contesti epidemiologici differenti da quello italiano.
Il presente studio va tuttavia considerato solo il primo passo di una ricerca più ampia volta a chiarire i molti interrogativi che tuttora permangono su questo lineage emergente, a partire dal suo effettivo impatto clinico ed economico e dalla possibile circolazione in categorie produttive diverse dalle ovaiole.
Clicca qui per leggere l’articolo completo.
Per tutti gli atti del convegno consultare www.patologiaviare.org/eventi/atti-dei-convegni/