

I reovirus aviari (ARV) sono la causa principale di artrite virale e tenosinovite in polli e tacchini e comportano notevoli perdite economiche per gli allevatori che si concretizzano in un peggior indice di conversione, minore uniformità e più scarti.
I reovirus aviari sono virus privi di involucro. Il loro materiale genetico è formato da 10 segmenti di RNA a doppia elica. Tali caratteristiche consentono un elevato livello di mutazione e ricombinazione, sfuggendo così all’immunità data dalla vaccinazione. I ceppi vaccinali classici, come S1133, 1733 e 2408 sono in uso dagli anni ’70, ma le omologie molecolari delle varianti a questi vaccini spesso sono inferiori all’80%.
Per anni, le varianti molecolari sono state rilevate e classificate in 5 e 6 cluster genotipici sulla base del gene S1, che codifica la proteina Sigma C. Tuttavia, sappiamo che le caratterizzazioni basate su S1 non forniscono il quadro completo e non si correlano con l’antigenicità, mentre la patogenicità di questi ceppi consente un metodo veloce e semplice per raggruppare gli isolati. Dato che i virus incorporati nei vaccini autogeni sono in parte selezionati sulla base della caratterizzazione di S1, è necessario riconsiderare questo approccio. Studi precedenti di laboratorio hanno suggerito come anche altri geni, quali M2 e L3 codificanti le proteine strutturali esterne, abbiano un’elevata variabilità e potrebbero essere buoni candidati per la classificazione molecolare delle varianti dei reovirus aviari; ciò potrebbe quindi essere associato anche alla patogenicità e antigenicità.
Dal 2015 è stata avviata una caratterizzazione molecolare basata sul profilo di S1 e classificazione degli isolati ottenuti dai laboratori californiani. Inoltre, sono stati selezionati isolati distinti per attuare una completa sequenziazione genomica e rilevare le differenze e variabilità dei diversi geni di reovirus.
Materiali e metodi
150 isolati di reovirus sono stati selezionati sulla base dell’importanza clinica, dell’anno di isolamento, del tessuto di provenienza e dell’effetto citopatico su coltura cellulare (CPE). Gli isolati confermati sono stati sottoposti a sequenziazione del gene S1 con RT PCR usando i primer inversi. Le sequenze derivanti sono state poi confrontate con i vaccini commerciali S1133, onde paragonare le omologie. Sono stati poi sviluppati gli alberi filogenetici per visualizzare la classificazione molecolare delle varianti dei reovirus aviari. 17 isolati rappresentativi sono quindi stati selezionati e il loro intero genoma sequenziato e classificato per controllare l’associazione dei geni. In seguito è stata eseguita un’analisi statistica e grafica con GraphPad Prisma.

Risultati
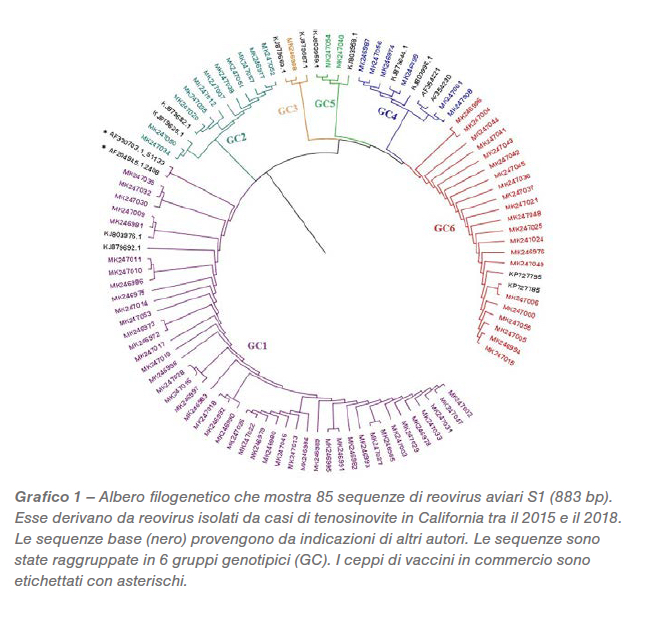
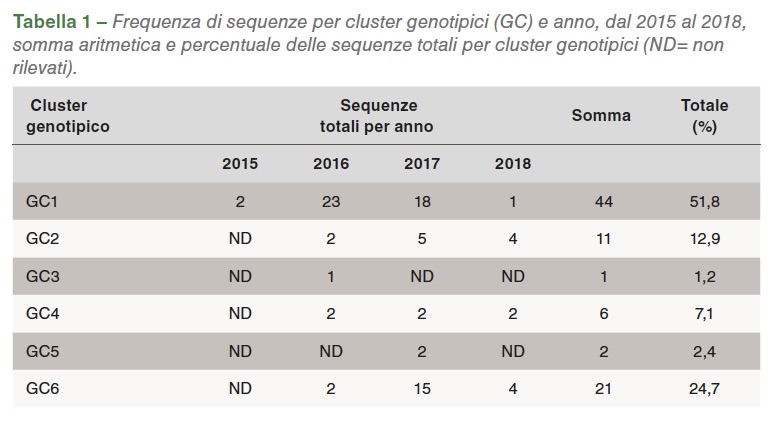
I segmenti S1 (1,088 bp) di 85 su 150 (56%) dei reovirus aviari sono stati amplificati con RT-PCR, poi sequenziati e classificati in 6 cluster genotipici (GC). Nel 2016 i ceppi GC1 erano predominanti, in circa l’80% delle varianti. Nel 2017 GC6 aumentava la propria quota fino al 31% delle sequenze, mentre GC1 restava il primo col 40% di isolamenti (Grafico 1). L’omologia media di ciascun GC rispetto a S1133 è raffigurata nella Tabella 1.
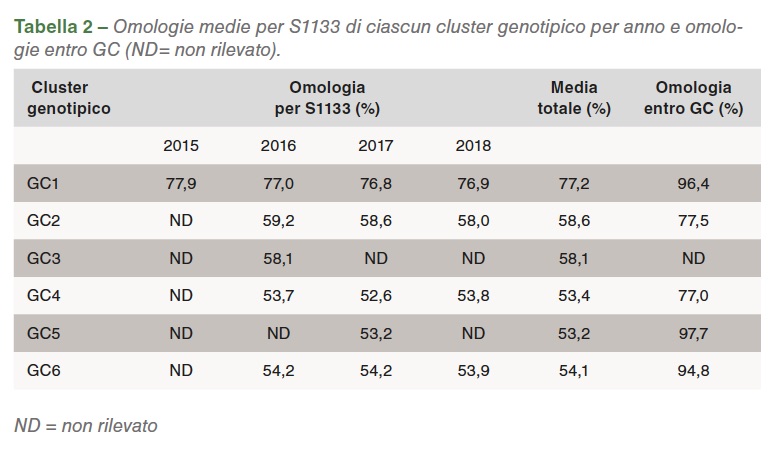
La percentuale di omologia rispetto a S1133 delle sequenze S1 per ogni anno è stata consistente: GC1 è stato il gruppo con l’omologia maggiore rispetto a S1133, mentre i gruppi GC4, GC5 e GC6 presentavano quella minore. Per stabilire la similarità delle sequenze S1 entro ciascun gruppo, è stata calcolata l’omologia dei vari cluster GC. Da alta a bassa, le omologie sono state: GC1 (96,4%), GC2 (77,5%), GC4 (77,0%), GC5 (97,7%) and GC6 (94,8%), vedi Tabella 2.

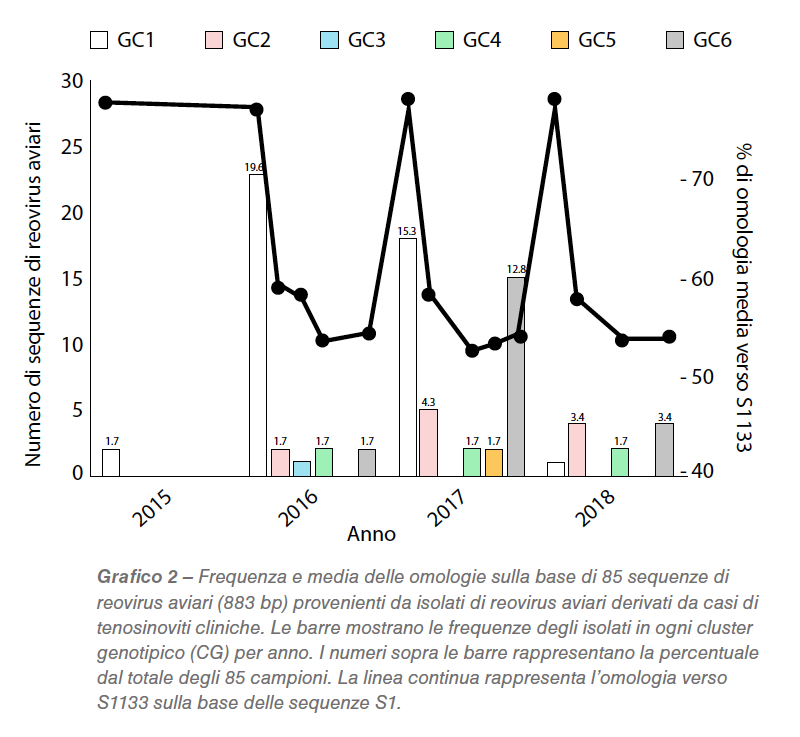
Nel Grafico 2 viene presentato un riassunto della distribuzione delle sequenze in diversi gruppi genetici e delle loro omologie per ciascun anno. Alla notevole riduzione delle sequenze raggruppate nel GC1 è seguito un aumento di GC2, GC4 e GC6. GC3 è stato identificato per la prima volta nel 2016, mentre GC5 nel 2017. 17 isolati associati a forma clinica in campo sono stati selezionati per la completa sequenziazione genomica e sierotipica.

Discussione
La sorveglianza molecolare è fondamentale per controllare e prevenire gli episodi di reovirus aviari patogeni. Con i primer descritti da Kant et al., solo 85 su 150 (56,6%) dei geni S1 sono stati rilevati, amplificati e sequenziati. La mancata amplificazione di oltre il 30% dei ceppi deriva parzialmente da divergenze molecolari di altre varianti di reovirus aviari. Kant et al., con gli stessi primer, dal 1980 al 2000, hanno sempre rilevato una quota di amplificazione leggermente maggiore (70%).
I metodi di caratterizzazione parziale del gene S1 hanno classificato i ceppi di reovirus aviari in 5 e 6 gruppi genotipici. La presente ricerca ha dimostrato che i ceppi isolati in California appartengono ai sei distinti cluster genotipici. I gruppi predominanti sono stati GC1 (51,8%), seguito da GC6 (24,7%) e GC2 (12,9%). Risultati simili sono stati descritti anche in Europa. Lu et al. hanno notato che la maggior parte delle sequenze apparteneva a GC5, seguito da GC4 e GC1. Palomino et al. hanno rilevato un maggiore numero di GC5, seguiti da GC4 e GC1.
La nomenclatura cluster viene usata per paragonare i virus isolati in varie parti del Paese e del mondo, anche se si dovrebbe tenere conto della dimensione dei frammenti, del numero delle sequenze analizzate, delle sequenze di base selezionate e della soggettività dell’analisi. Questi fattori giocano tutti un ruolo nella conformazione dei cluster. Tra il 2015 e il 2018, la rappresentazione dei cluster degli isolati genotipici dei reovirus aviari in California è cambiata. Si è visto un calo di GC1 e un aumento di GC6 (Tabella 2).
INSERIRE TABELLA 2 E GRAFICO 2
Sono diversi i fattori che potrebbero avere influenzato questo cambiamento, incluso l’utilizzo di vaccini autogeni. L’uso di certi GC come antigeni predominanti nei vaccini autogeni potrebbe aver avuto un ruolo rilevante nel cambiamento della rappresentazione dei cluster genetici. L’ipotesi in campo è che i vaccini non omologhi e inattivati offrano una protezione parziale verso le infezioni di campo senza eliminare l’escrezione virale da parte dei soggetti infetti. Ciò consente la selezione di ceppi diversi da quelli vaccinali e altera la rappresentazione dei reovirus aviari nell’ambiente. Le omologie con vaccini commerciali di riferimento sono state maggiori nel gruppo GC1. Tuttavia, GC1, che è il gruppo presente nei vaccini come ceppo, ha mostrato un’omologia inferiore al 78%, mentre gli altri gruppi GC si attestavano su una media del 53,2% per S1133, quindi assai distante dai virus usati nei vaccini commerciali vivi e inattivati. Ciò spiega la mancanza di efficacia di questi vaccini nel generare la protezione attesa. Sulla base delle omologie rilevate negli anni, si nota che ciascuno dei cluster ha mantenuto il proprio livello di omologia rispetto a S1133 dal 2016 (Grafico 2).
La bibliografia è disponibile su richiesta
Dagli atti della 68esima Western Poultry Disease Conference









